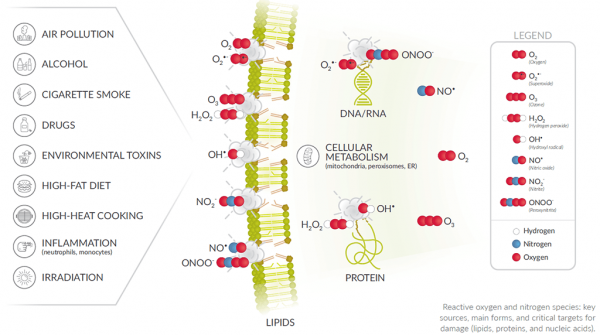
Oxidativer Stress kann durch zwei Methoden bestimmt werden. Einmal durch die direkte Messung der reaktiven Sauerstoffspezies (ROS) oder durch die indirekte Messung der Schädigungen von Lipiden, Proteinen und Nukleinsäuren. Diese Schädigungen werden durch eine Überproduktion der ROS verursacht. Obwohl die direkte Messung von ROS genauer ist, werden die indirekten Methoden aufgrund der relativen Stabilität von Schadensmarkern an Biomolekülen öfters eingesetzt. In diesem Blog finden Sie eine Aufschlüsselung verschiedener Assays, die zum Nachweis der häufigsten Biomarker für oxidative Schäden verwendet werden.
ROS
Sauerstoff wird als Teil des normalen Stoffwechsels reduziert, was zur Bildung verschiedener ROS, darunter Wasserstoffperoxid (H2O2) und Superoxid (O2•−) , führt. Schäden an zellulären Makromolekülen entstehen, wenn es zu unkontrollierten Oxidationen im biologischen System kommt. Assays für ROS erkennen nicht die Quelle der ROS-Produktion. Wenn das Versuchsmodell jedoch unter Stress steht, ist ein Anstieg der ROS und die Veränderung der molekularen Komponenten wahrscheinlich.
H2O2
H2O2 kann mit empfindlichen Sonden wie ADHP nachgewiesen werden, die an ein Enzym wie HRP gekoppelt sind. Die Assay-Spezifität wird signifikant verbessert, wenn ein H2O2 -Binder, wie z.B. eine Katalase, als Kontrolle einbezogen wird.1
Dihydroethidium
Dihydroethidium (Hydroethidin oder DHE) kann direkt in lebenden Zellen eingesetzt werden. Diese redoxempfindliche Sonde wird durch O2•− zur Bildung von 2-Hydroxyethidium (Ex: 500-530 nm, Em: 590-620 nm), oder durch unspezifische Oxidation durch H2O2, oder andere ROS-Quellen zur Bildung von Ethidium (Ex: 480 nm, Em: 576 nm) oxidiert.2,3
Xanthinoxidase
Xanthinoxidase (XO) produziert sowohl H2O2 als auch O2•−.1 Die Aktivität dieses Enzyms kann gemessen werden, indem das Nebenprodukt (H2O2) des Abbaus von Hypoxanthin durch XO über eine Sonde erfasst wird. 2,3
RNS
Reaktive Stickstoffspezies (RNS) entstehen ebenso bei oxidativem Stress. Hohe Mengen an Stickstoffmonoxid (NO•), synthetisiert durch die Stickstoffmonoxid-Synthase (NOS) und O2•− führen zur Bildung von Peroxynitrit. NO• selbst reagiert mit Thiolen und Eisen-Schwefel-Enzymen, während Peroxynitrit mit Tyrosinresten zu Nitrotyrosin reagiert.4,5
Nitrat (NO3−)/Nitrit (NO2−)
NO3− und NO2− sind Produkte der in vivo NO•-Reaktionen, deren Gesamtproduktion entweder mit Griess-Reagenzien oder DAN nachgewiesen werden kann.4,5 Diese Assays konvertieren zunächst NO3− zu NO2− unter Verwendung von NADPH-abhängiger Nitratreduktase. Mittels anschließender Reaktion mit Griess-Reagenzien oder DAN kann die Gesamtkonzentration von NO2−-Ionen bestimmt werden..
NOS
Die NOS-Aktivität wird in Geweben und Zellen nachgewiesen. Dazu wird die von NOS angeregte Umwandlung eines radioaktiv markierten Arginins in Citrullin gemessen. Alternativ kann die in vitro NOS-Aktivität anhand der Griess-Reaktion nachgewiesen werden. Hierbei wird überschüssiges NADPH (zugegeben als Co-Faktor der NOS-Aktivität) mit einem Oxidationsschritt, katalysiert durch die Laktatdehydrogenase, entfernt.
DNA/RNA-Schäden
Guanin ist die Base, die am anfälligsten für Oxidation ist, wenn DNA und RNA beschädigt werden. Die Reparaturprozesse, die eingeleitet werden, um diesen Schaden zu beheben, setzen die folgenden oxidierten Guanin-Arten in den Urin frei:
- 8-Hydroxyguanin — Eine ribose-freie Base
- 8-Hydroxyguanosin — Ein Nukleosid aus der RNA
- 8-Hydroxy-2’-deoxyguanosin — Ein Deoxynukleosid aus der DNA
Assays, die DNA/RNA - Schäden nachweisen, können in zwei Kategorien eingeteilt werden. Zu der ersten Kateogorie gehören Assays, die mehrere oxidierte Guanin-Spezies nachweisen können. Sie erfassen einen vollständigeren Satz biologisch relevanter Produkte mit oxidativer Schädigung. Zu der zweiten Kategorie zählen Assays, die sich auf die Analyse von nur einem Molekül (z.B. 8-Hydroxy-2'-desoxyguanosin) beschränken.6-9

Abbildung 1: DNA/RNA-Schäden. . Beachten Sie, dass die O2• und ONOO- Moleküle beispielhafte ROS- und RNS-Spezies sind, die Schäden verursachen. Es gibt viele weitere Moleküle, die auch DNA und/oder RNA schädigen können .
Proteinoxidation und Nitrierung
Der häufigste Marker für Proteinoxidation ist der Gehalt an Proteincarbonyl.10 Diese Kationen binden an Proteine und führen in Verbindung mit den ROS zur Bildung von Aminosäurederivaten mit Carbonylgruppen (Ketone, Aldehyde). Zigarettenrauch und Aldehyde führen u.a. auch zu Carbonylverbindungen in Proteinen. Alternativ erzeugt die ROS-Exposition an Methioninresten eines Proteins Proteinmethioninsulfoxid (MetO). Diese oxidative Modifikation wird, wenn nicht durch MetO-Reduktasen rückgängig gemacht, weiter zu Methioninsulfon oxidiert und kann zu Proteinfehlfunktionen führen. Das Vorhandensein von Nitrotyrosin auf Proteinen wird als Marker für Peroxynitrit verwendet, der in vivo gebildet wird, wenn NO• mit O2•− reagiert.12 Da Peroxynitrit eine heterolytische Spaltung erfährt, werden freie Nitroniumionen sowie Nitratprotein-Tyrosinreste gebildet. NO• kann Proteine durch S-Nitrosylation direkt modifizieren, wobei eine NO-Gruppe an Thiolgruppen von Cysteinresten bindet, was zur Bildung von S-NO führt.

Abbildung 2: Proteinschäden. Beachten Sie, dass H2O2 und OH• nur beispielhaft für Proteinchädigende ROS-Spezies sind. Es gibt viele weitere Moleküle, die auch Proteine beschädigen können.
Carbonylgehalt
Eine geeignete Technik zum Nachweis des Carbonylgehalts in Proteinpräparaten ist die Reaktion zwischen DNPH und Proteincarbonyl. Dabei entsteht eine Schiff'sche Base, die ein entsprechendes Hydrazin erzeugt, das spektrophotometrisch gemessen werden kann.
Methioninsulfoxid (MetO)
Ein für MetO spezifischer Antikörper wird verwendet, um oxidative Modifikationen zu beobachten, indem Proteine mit MetO-Resten nachgewiesen werden.11
Nitrotyrosin
Ein für Nitrotyrosin spezifischer Antikörper wird zum Nachweis der Nitrierung verwendet.
S-Nitrosylierung
Die Protein-S-Nitrosylierung kann mit einem Biotin-Switch-Assay direkt visualisiert werden. Dieses Verfahren spaltet S-NO-Bindungen (nach dem Blockieren vorhandener freier Thiole), um die nun freien Thiolgruppen zu biotinylieren.13
Lipidperoxidation
Die Lipidperoxidation führt zur Bildung von hochreaktiven und instabilen Hydroperoxiden aus ungesättigten Lipiden. Sie können direkt oder indirekt durch die verschiedenen Zersetzungsprodukte (z.B. Alkane, Ketone, Aldehyde) von instabilen Hydroperoxiden gemessen werden. 8-Isoprostan, das durch zufällige Oxidation von Gewebephospholipiden hergestellt wird, gilt derzeit als einer der zuverlässigsten Biomarker für die Lipidperoxidation in vivo.14 Es ist ein spezifisches Produkt der Lipidperoxidation, das stabil ist und in allen normalen biologischen Flüssigkeiten und Geweben in nachweisbaren Mengen vorhanden ist. Der Grad der Veresterungen in Phospholipiden kann verwendet werden, um das Ausmaß der Lipidperoxidation an den gewünschten Zielstellen zu bestimmen.

Abbildung 3: Lipischschaden. Beachten Sie, dass O2 und O2• − nur beispielhaft für Lipidschädigende ROS-Spezies sind. Es gibt viele weitere Moleküle, die auch Lipide beschädigen können.
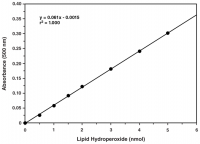
Lipidhydroperoxide (LPOs)
LPOs können effizient extrahiert und direkt gemessen werden, indem Redoxreaktionen mit Eisenionen eingesetzt, um den gesamten Hydroperoxidgehalt zu einem bestimmten Zeitpunkt zu ermitteln.
4-Hydroxynonenal (4-HNE)
4-HNE-Proteinaddukte sind in der Regel stabiler als MDA-Proteinaddukte. 1,4-Dihydroxynonan-Mercaptursäure (DHN-MA), der wichtigste Harnstoffmetabolit von 4-HNE, ist ein zusätzlicher Biomarker, der gemessen werden kann.
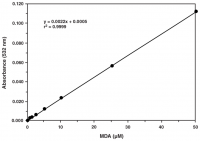
Malondialdehyd (MDA)
MDA-Assays verwenden eine Thiobarbitur-Reaktion und werden daher Thiobarbituric Acid Reactive Substances (TBARS) Assays genannt. Thiobarbitursäure reagiert mit verschiedenen Aldehyden, die bei der Lipidperoxidation zusätzlich zu MDA entstehen.15
8-lsoprostan
8-Isoprostan, das durch zufällige Oxidation von Gewebephospholipiden hergestellt wird, gilt derzeit als einer der zuverlässigsten Biomarker für die in vivo Lipidperoxidation.16 Es wird typischerweise entweder mit einem Immunassay oder LC-MS oder GC-MS analysiert.
Kit Empfehlungen
ROS Assays
| Produkt | Probentyp | Was wird gemessen | Methode |
| Hydrogen Peroxidas Cell-Based Assay Kit | Kultivierte Zellen | Extrazelluläres H2O2 | Fluoreszenzmessung auf einer Platte (Ex: 530-560 nm, Em: 590 nm) |
| Mitochondrial ROS Detection Assay Kit | Lebende Zellen | Mitochondriale ROS | Fluoreszenzmessung auf einer Platte (Ex: 480-515 nm, Em: 560-600 nm), oder Durchflusszytometrie |
| ROS Detection Cell-Based Kit (DHE) | Lebende Zellen | ROS | Fluoreszenzmessung auf einer Platte (Ex: 480-520 nm, Em: 570-600 nm), oder Durchflusszytometrie |
| Xanthine Oxidase Fluorometric Assay Kit | Plasma, Serum und Gewebehomogenate | XO Aktivität | Fluoreszenzmessung auf einer Platte (Ex: 520-550 nm, Em: 585-595 nm), oder Durchflusszytometrie |
RNS Assays
| Produkt | Probentyp | Was wird gemessen | Methode |
| Nitrate/Nitrite Colorimetric Assay Kit | Plasma, Serum, Urin, Gewebekulturmedien und Gewebehomogenate | NO• Metaboliten | Farbmetrische Messung auf einer Platte (540-560 nm) |
| Nitrate/Nitrite Colorimetric Assay Kit (LDH method) | Plasma, Serum, Urin und Gewebehomogenate | In vitro NOS Aktivität und NO• Metaboliten | Farbmetrische Messung auf einer Platte (530-550 nm) |
| Nitrate/Nitrite Fluorometric Assay Kit | Plasma, Serum, Urin, Gewebekulturmedien und Gewebehomogenate | NO• Metaboliten | Fluoreszenzmessung auf einer Platte (Ex: 360-365 nm, Em: 430 nm) |
| NOS activity Assay Kit | Zelllysate und aufgereinigte Präparate | NOS Aktivität | Flüssigzintillationszählung |
DNA/RNA Damage Assays
| Produkt | Probentyp | Was wird gemessen | Methode |
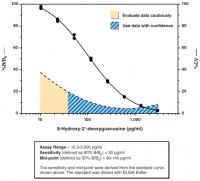
| DNA/RNA Oxidative Damage (Clone 7E6.9) ELISA Kit | Urin (andere Poben wurden noch nicht validiert, können aber vielleicht genutzt werden) | 8-Hydroxy-2'-deoxyguanosin und 8-Hydroxyguanosin | Farbmetrische Messung auf einer Platte (405-420 nm) |
| DNA/RNA Oxidative Damage (High Sensitivity) ELISA Kit | Urine, Zellkulturmedium, Zelllysate, Gewebsproben, Speichel, Plasma oder Serum Proben | 8-Hydroxy-2'-deoxyguanosin, 8-Hydroxyguanosin and 8-Hydroxyguanine | Farbmetrische Messung auf einer Platte (405-420 nm) |
Protein Oxidation and Nitration Assays
| Produkt | Probentyp | Was wird gemessen | Methode |
| Protein Carbonyl Colorimetric Assay Kit | Plasma, Serum, Urin, Gewebehomogenate und Zelllysate | Protein-Carbonylgehalt | Farbmetrische Messung auf einer Platte (360-385 nm) |
| Methionine Sulfoxide Immunoblotting Kit | Plasma, CSF, Zell/Gewebs-Lysate oder semi-reine/aufgereinigte Proteine | Proteine die MetO-Reste beinhalten | Immunochemische Detektion mittels Westernblot |
| Nitrotyrosine IP Kit | Zelllysate | Nitrierter Tyrosingehalt | Affinität-Sorbens-Erfassung und Elution mittels Westernblot oder Proteomanalysen |
| S-Nitrosylated Protein Detection Kit (Biotin Switch) | Ganze Zellen oder Gewebe | S-NO Proteine | Streptavidin-basierte Detektion mittels Westernblot oder Fluoreszenzmikroskopie |
| S-Glutathionylated Protein Detection Kit | Ganze (permeabilisierte) Zellen | Protein-PSSG adukte | Streptavidin-basierte Detektion mittels Durchflusszytometrie oder Fluoreszenzmikroskopie, oder IP/Avidin-Überlappungsanalyse |
Lipid Peroxidation
| Produkt | Probentyp | Was wird gemessen | Methode |
| DHN-MA EIA Kit | Urin | DHN-MA, a 4-HNE Metabolite | Farbmetrische Messung auf einer Platte (405-414 nm) |
| 8-Isoprostane ELISA Kit | Plasma, Serum, Urin, Lavageflüssigkeiten, Gewebehomogenate und Kulturmedium | 8-Isoprostan | Farbmetrische Messung auf einer Platte (405-420 nm) |
| 8-Isoprostane Express ELISA Kit | Plasma, Serum, Urin, Lavageflüssigkeiten, Gewebehomogenate und Kulturmedium | 8-Isoprostan | Farbmetrische Messung auf einer Platte (405-420 nm) |
| STAT-8-Isoprostane ELISA Kit | Plasma, Serum, Urin, Lavageflüssigkeiten, Gewebehomogenate und Kulturmedium | 8-Isoprostan | Farbmetrische Messung auf einer Platte (405-420 nm) |
| Liquid hydroperoxide (LPO) Assay Kit | Gewebe, kultivierte Zellen, Pflanzenmaterial, Nahrung und biologische Flüssigkeiten | LPOs | Farbmetrische Messung auf einer Platte (500 nm) |
| Liquid hydroperoxide (LPO) Assay Kit (96 well) | Gewebe, kultivierte Zellen, Pflanzenmaterial, Nahrung und biologische Flüssigkeiten | LPOs | Farbmetrische Messung auf einer Platte (500 nm) |
| TBARS Assay Kit | Plasma, Serum, Urin, Gewebehomogenate und Zelllysate | MDA-TBA Addukt | Farbmetrische Messung auf einer Platte (530-540 nm) oder Fluoreszenzmessung (ex 530 nm, em 550 nm) |
| TBARS (TCA Method) Assay Kit | Plasma, Serum, Urin, Gewebehomogenate und Zelllysate | MDA-TBA Addukt | Farbmetrische Messung auf einer Platte (530-540 nm) oder Fluoreszenzmessung (Ex: 530 nm, Em: 550 nm) |
Referenzen
- Kelley, E.E., Khoo, N.K., Hundley, N.J., et al. Free Radic. Biol. Med. 48(4), 493-498 (2010).
- Michalski, R., Michalowski, B., Sikora, A., et al. Free Redic. Biol. Med. 67, 278-284 (2014).
- Zhao, H., Kalivendi, S., Zhang, H., et al. Free Radic. Biol. Med.34(11), 1359-1368 (2003).
- Green, L.C. Wagner, D.A., Glogowski, J., et al. Anal. Biochem.126, 131-138 (1982).
- Misko, T.P., Schilling, R.J., Salvemini, D., et al. Anal. Biochem. 214, 11-16 (1993).
- Gan, W., Nie, B., Shi, F., et al. Free Radic. Biol. Med.52, 1700-1707 (2012).
- Shi, F., Nie, B., Gan, W., et al. Free Radic. Res.46(9), 1093-1098 (2012).
- Roszkowski, K. and Olinkski, R. Cancer Epidemiol. Biomarkers Prev.21, 629-634 (2012).
- Weimann, A., Broedbaek, K., Henriksen, T., et al. Free Radic. Res.46(4), 531-540 (2012).
- Stadtman, E.R. and Oliver, C.N. J. Biol. Chem.266(40), 2005-2008 (1991).
- Oien, D.B., Canello, T., Gabizon, R., et al. Arch. Biochem. Biophys.485, 35-40 (2009).
- Beckman, J.S. and Koppenol, W.H. Am. J. Physiol.271, C1424-C1437 (1996).
- Jaffrey, S.R. and Snyder, S.H. Sci STKE2001(86), pl1 (2001).
- Esterbauer, H., Schaur, R. J., and Zoliner, H. Free Radic. Biol. Med.11, 81-128 (1991).
- Armstrong, D. and Browne, R. Free Radicals in Diagnostic Medicine366, 43-58 (1994).
- Roberts, L.J. and Morrow, J.D. Free Radic. Biol. Med.28, 505–513 (2000).